[Paper Review] Subject-Aware Contrastive Learning for Biosignals
본문은 Constrastive learning 기반 Self-supervised leanring을 활용하였습니다.
Overview
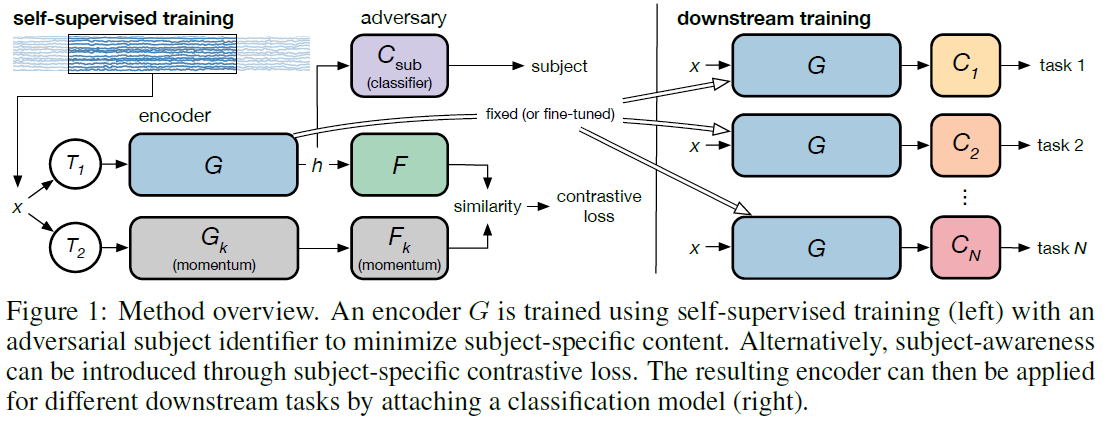
네트워크 구조
Abstract
Biosignals의 한계
Datasets for biosignals, such as electroencephalogram (EEG) and electrocardiogram(ECG), often have noisy labels and have limited number of subjects (<100).
- subject-invariance loss: improves representation quality for these tasks
- subject-specific loss: increases performance when fine-tuning with supervised labels
Introduction
기여도
- Apply self-supervised learning to biosignals
- Develop data augmentation techniques for biosignals: In this work, we develop domain-inspired augmentation techniques. For example, the power in certain EEG frequency bands has been shown to be highly correlated with different brain activities. Thus, we use frequency-based perturbations to augment the signal.
- Integrate subject awareness into the self-supervised learning framework
Temporal specific transformations(cutout, delay)가 representation을 학습하는 데 가장 효과적이었으며, 그 뒤로는 signal mixing, sensor perturvations (dropout and cutout), 그리고 bandstop filtering이었습니다.
Methods
Self-supervised learning은 데이터 변형 기술과 대조 학습을 사용하여 수행됩니다. 먼저, 대조 학습 프레임워크는 사람 간 가변성을 해결하기 위해 사람 인식(subject awareness)을 위한 layer가 추가되었습니다. 다음으로, 생체 신호 표현을 보다 효과적으로 학습하기 위해 특정 변형 기술을 설계하고 도입하였습니다. 마지막으로 제안된 접근 방식은 다음 섹션에서 설명하는 다운스트림 작업에 적용됩니다.
Model and Contrastive Loss function

네트워크 구조
Encoder \(G(T(x_i))\)는 변형 기법 \(T_1\)을 사용하여 데이터 \(x_i\)를 latent represenation \(h_i\)로 인코딩합니다. Self-supervised training 구간에서 \(h_i\)는 모델 \(F(h_i)\)를 통해 output \(q_i\)로 나옵니다. G 및 F의 사본인 \(G_k\)와 \(F_k\)는 변형 기법 \(T_2\)를 통해 \(x_i\)에서 \(k_i\)를 생성하는 데 사용됩니다. 모델 \(G_k\) 및 \(F_k\)는 momentum으로 업데이트됩니다. 또한, \(q_i\) 및 \(k_i\)는 L2-norm으로 정규화(normalization)되었습니다. \[\begin{gather} q_i = F(G(T_1(x_i))) \\ k_i = F_k(G_k(T_2(x_i))) \end{gather}\]
모델은 \(T_1(x_i)와 T_2(x_i)\) 간의 상호정보를 최대화하도록 학습됩니다. 상호 정보 최대화는 InfoNCE로 추정됩니다. \[l_i = - log \frac{exp({q_i}^T k_i / \tau)}{\sum_{j=0}^N exp({q_i}^T k_i / \tau)}, \tag{1}\]
- \({q_i}^T k_i\): \(q_i\)와 \(k_i\)의 내적(inner product)로 similarity metric으로 사용
- \(\tau\): learnable temperature parameter
In Eq. 1, qT i ki is contrasted against the inner product of qi and N 1 negative examples. The momentum update of Gk and Fk enables the use of negative examples from previous batches to increase the number of negative examples [12]. \[l_{sub, i} = - \sum_{j=0}^{N_{sub}-1} 1_{j=s_i} log C_{sub}^j(G(x_i)), \tag{2}\]
- \(N_{sub}\): 피험자의 수
- \(s_i\): 샘플 \(i\)의 피험자 번호
Experiments and Results
EEG: PhysioNet Motor Imagery Dataset
- Subjects: 106 명 (3명 데이터 제외)
- Class: 4 개(closing the right fist, closing the left fist, closing both fists, and moving both feet)
- Time: 4 sec
- Channels: 64 개
- Sampling rate: 160 Hz
- Preprocessing:
- Re-referening: channel average
- Normalization: mean and standard deviation
Experimental Setup.
Encoder G:
- 90 명의 데이터를 사용하여 self-supervised learing
- Input은 4 초의 시간 중에서 랜덤으로 2초를 선택하여 320 samples을 사용함
- 256-dimensional embedding vector
- Batch size: 400, steps: 270000
- 모델 끝에 logistic-regression lienar classifier를 추가하여 90 명의 데이터에 대한 정확도를 평가함
- 데이터는 cue 다음 0.25초 이후의 신호를 사용함
- Intersubject testing을 위해 나머지 16명의 데이터에 평가함
Model F:
Downstream taks + classifier:
- 2-class: rigth fist, left fist
- 4-class: right fist, left fist, both fists, both feet
- Setting:
- Learning rate: 1e-3
- Batch size: 256
- Epochs: 2000
Impact of Transformation.
Impact of Subject-Aware Training.
- No augmentation
- Base SSL
- Subject-specific
- Subject-invariant
- Random encoder: Randomly-initialized encoder was used as a baseline for comparison